La recente disponibilità di strumenti molecolari innovativi permette di capire e cogliere meglio le effettive differenze tra razze e sub-specie. Al di là delle marcate differenze tra bufalo swamp e river, la genomica ci conferma che la Bufala Mediterranea Italiana appartiene ad un gruppo separato e ben definito che la differenzia anche dalle altre razze presenti nel continente europeo, come la bufala Ungherese o Rumena. Queste conoscenze vanno a tutto vantaggio della selezione e del corretto uso dei riproduttori per il miglioramento della BMI. Una breve raccolta dei lavori più recenti pubblicati su riviste scientifiche internazionali e riguardanti la specie bufalina.
Bufalo d’acqua
Il bufalo d’acqua comprende due sottospecie: il bufalo di fiume (Bubalus bubalis bubalis) ed il bufalo di palude (Bubalus bubalis carabanensis). Le due sottospecie sono geneticamente distinte con un diverso numero di cromosomi (50 e 48, rispettivamente), e con caratteristiche differenti in termini di morfologia, fisiologia, produzione e riproduzione (Colli et al., 2018, Minervino et al., 2020).
Il bufalo di palude ha un fenotipo piuttosto omogeneo, sebbene esista una marcata variabilità legata alla posizione geografica. Nonostante questo, non è possibile identificare delle vere e proprie razze. Al contrario, il bufalo di fiume comprende diverse razze ben identificate. Secondo un rapporto dell’Organizzazione delle Nazioni Unite per l’alimentazione e l’agricoltura (FAO) basato sui dati nazionali, ci sono 123 razze di bufali, 90 nella sola Asia (FAO). Esiste molta variabilità nei caratteri produttivi e fenotipici ed uno di essi, la conformazione delle corna, può essere utilizzato in alcuni casi come marker fenotipico di purezza raziale. La Figura 1 mostra alcuni esemplari delle due sub-specie.
Figura 1 – Razze di bufali: A) Carabao – Filippine; B) Fuling – Cina; C) Murrah – Brasile; D) Bufala Mediterranea Italiana.
Gli animali di tipo mediterraneo hanno il mantello nero (come la Bufala Mediterranea Italiana), marrone o grigio oscuro. Le loro corna sono piatte nella parte inferiore e sono rivolte all’indietro, mentre le punte sono rivolte verso l’alto e all’interno. Il Mediterraneo è una razza compatta con un petto ampio e profondo, ma dorso e groppa corte. La mammella è di media grandezza con quarti e metà perpendicolari e con capezzoli cilindrici (Moioli and Borghese, 2005, Borghese and Moioli, 2016).
Minervino et al., (2020), riportano una tabella con l’elenco delle razze più importanti e popolazioni riconosciute di bufalo d’acqua. La Bufala Mediterranea Italiana (BMI) viene classificata individualmente rispetto al resto delle razze mediterranee, essendo principalmente selezionata per mozzarella ed industria di trasformazione.
Esistono tre principali razze di bufalo di fiume distribuite in tutto il mondo, soprattutto per il loro potenziale genetico per la produzione di latte, ovvero: Murrah, Nili-Ravi e il Mediterraneo italiano (Borghese and Moioli, 2016). La Murrah è la razza di bufali più rappresentata e conosciuta al mondo, essendo stata esportata in diversi paesi, come Brasile, Asia orientale e Bulgaria (Moioli and Borghese, 2005, Yadav et al., 2017). In quest’ultimo paese esiste un ampia popolazione originata dall’incrocio tra il Murrah indiano e la razza mediterranea locale. Molti animali sono stati esportati dalla Bulgaria nei paesi vicini, come la Romania e la Germania, ma anche in Brasile e Venezuela. La seconda razza più rappresentata al mondo è la Nili-Ravi, maggiormente rappresentata in Asia dove è principalmente allevata per il latte. La terza razza è il Bufalo Mediterraneo, discendente dal bufalo di fiume introdotto in Europa dall’India durante l’occupazione araba dell’VIII secolo in Sicilia e nell’Italia meridionale (Minervino et al., 2020). La Bufala italiana ha un’elevata resa e qualità del latte, ed è la più selezionata dal punto di vista genetico per la produzione di latte diretto all’industria casearia, con un libro genealogico istituito nel 1980, e l’Associazione Nazionale Allevatori Specie Bufalina (ANASB) istituita nel 1979.
Dati attuali sulla popolazione di bufali nel mondo (Figure 2), mostrano come nel continente Europeo la razza BMI ripresenti l’87% della popolazione, seguita dalla Romania e Bulgaria solo con il 3% (Minervino et al., 2020).
Figura 2 – Distribuzione della popolazione di bufali nei vari paesi del mondo. La legenda indica il numero di capi in ogni paese; t, mille; m, milioni.
L’allevamento del bufalo è di grande attualità
Rispetto ad altri ruminanti, la bufala ha una maggiore capacità di convertire alimenti a basso valore nutritivo ed un’elevata capacità di adattamento e sopravvivenza in diversi ambienti con differenti topografia, clima e vegetazione (Li et al., 2020). In particolare, per quanto riguarda la sua tolleranza al caldo, recenti studi molecolari hanno dimostrato che le razze di bufali storicamente allevate in un clima caldo possono aver sviluppato una maggiore tolleranza al calore (Mokhber et al., 2019). A questo si aggiunge, inoltre, che i bufali hanno una longevità maggiore dei bovini (possono superare i 30 anni, mantenendo la loro capacità riproduttiva fino a 18-25 anni).
Diversità e struttura genetica
Al di là delle caratteristiche fenotipiche che differenziano le varie razze o sub-specie, uno degli obiettivi di tutti i ricercatori a livello mondiale è sempre stato quello di capire e studiare cosa si “nasconde” dietro al fenotipo, e quindi la struttura genetica. È la struttura genetica che ci permette di capire davvero quanto 2 individui siano simili e cosa determina, ad esempio, la loro produzione. L’ultimo decennio è stato sicuramente prodigo di novità ed ha fornito strumenti innovativi (come ad esempio pannelli di marcatori ad alta densità) che hanno permesso di esplorare aspetti sino ad ora meno conosciuti.
Alla luce delle nuove tecnologie di sequenziamento di ultima generazione e, vista la notevole importanza economica che il bufalo riveste in Italia e nel mondo, è partita, nel 2010, un’iniziativa per sequenziare e caratterizzare il genoma bufalino, promossa dal Parco Tecnologico Padano di Lodi con la supervisione dell’Associazione Nazionale Allevatori della Specie Bufalina (ANASB).
La sequenza completa del genoma bufalino è stata completata nel 2013 per la Bufala Mediterranea Italiana da parte del Water Buffalo Genome Consortium. Il processo di annotazione, cioè quel lavoro che progressivamente colloca i diversi geni nella posizione precisa in cui si trovano sui diversi cromosomi, è tutt’ora in corso. Il sequenziamento costituisce il punto di partenza per tutti gli studi genetici e di comparazione sulle diverse razze bufaline e per avere accesso ai più moderni strumenti per lo sviluppo del potenziale genetico della specie.
Nel 2017 il proseguimento del lavoro cominciato nel 2013 (Iamartino et al., 2017) con la collaborazione di molti istituti di ricerca, unitamente al contributo di ANASB e del laboratorio genetica e servizi di AIA, ha contribuito allo sviluppo e alla validazione di un chip a DNA di circa 90.000 marcatori da utilizzare per le analisi genomiche delle razze bufaline di tipo River, a cui appartiene la Bufala Mediterranea. Il chip “90K Affimetrix” costituisce, di fatto, una buona base di partenza per l’avvio di programmi di selezione genomica e lo studio della biodiversità, soprattutto nelle popolazioni bufaline di tipo River. Inoltre, la disponibilità di questo chip consente oggi di poter gettare le basi della selezione genomica nella Bufala Mediterranea.
Infatti, utilizzando questi strumenti innovativi sono stati pubblicati successivamente diversi studi sulla struttura e la diversità genetica del bufalo. In questa breve revisione, cercheremo di sintetizzare alcuni dei principali risultati pubblicati riguardanti la Bufala Mediterranea Italiana ed il suo rapporto con altri tipi genetici bufalini.
Nel 2018, Colli et al., (2018) pubblicano il primo studio condotto con SNP, comprendente diverse razze di bufali. Nel lavoro, dal titolo “New Insights on Water Buffalo Genomic Diversity and Post-Domestication Migration Routes From Medium Density SNP Chip Data”, vengono sia stimati i livelli di diversità molecolare che descritta la struttura della popolazione, permettendo di rilevare relazioni storiche tra popolazioni ed eventi migratori.
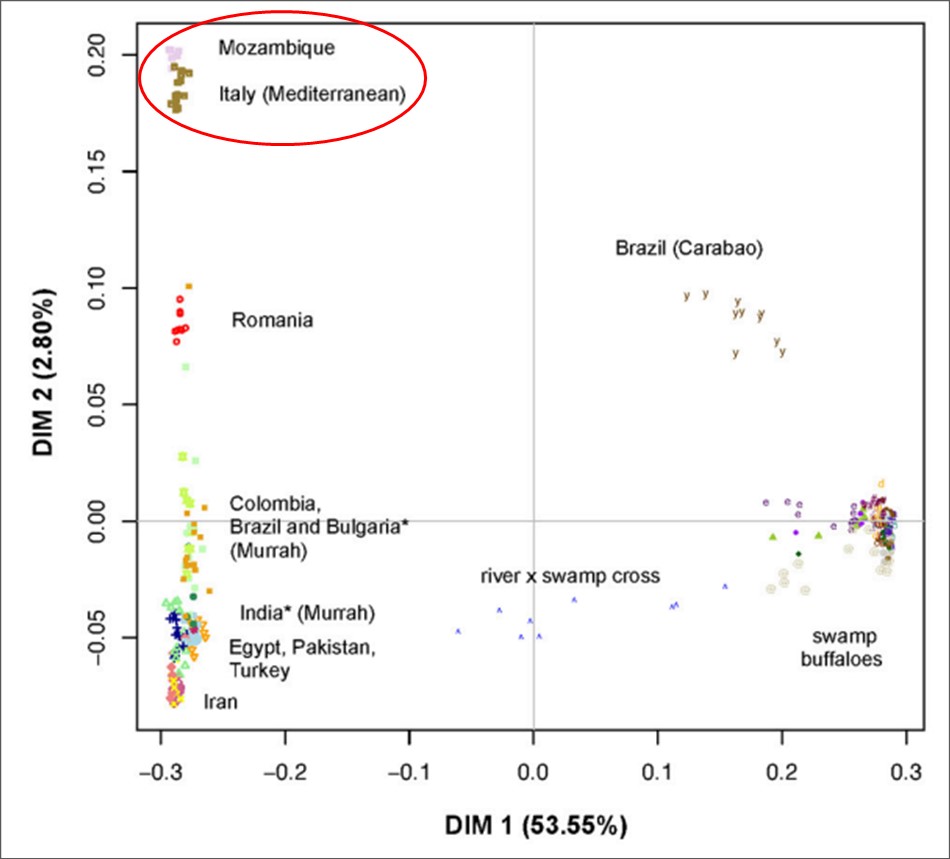
Uno dei risultati più interessanti ed immediati può essere osservato nella la Figura 3 che mostra, attraverso un’analisi detta in gergo multivariata, le relazioni tra le differenti popolazioni mondiali. Più in dettaglio, si possono identificare diversi gruppi: (i) un primo gruppo racchiude la Bufala Italiana con gli animali provenienti dal Mozambico, (ii) un secondo gli animali provenienti dalla Romania, che formano un gruppo separato da tutte le altre popolazioni, (iii) un terzo gruppo comprendente le popolazioni Murrah provenienti da Bulgaria, Brasile, India, e Colombia; iv) animali provenienti da Turchia, Egitto e Pakistan e per ultimo v) le popolazioni dall’Iran.
Figura 3 – Grafico MDS (multidimensional scale plot) che rappresenta attraverso procedimento multifattoriale il posizionamento delle diverse razze di bufalo mondiali.
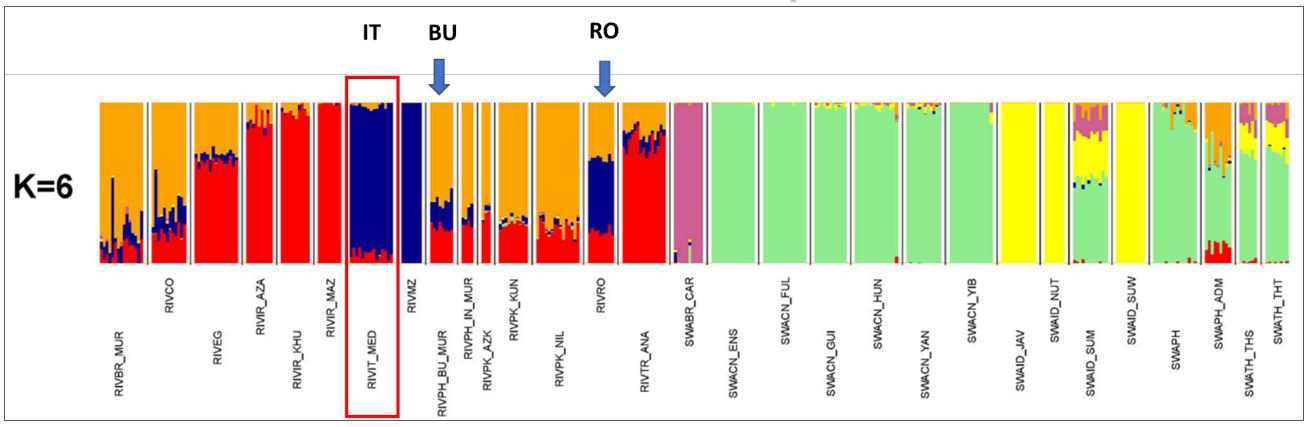
Attraverso metodologie più avanzate (approcci Bayesiani e admixture) è possibile assegnare ogni individuo ad un cluster, permettendo quindi di visualizzare quali componenti genetiche caratterizzano una determinata razza e anche di stabilire la storia della sua formazione. Nella figura riportata in basso si può vedere il risultato emesso dal software per la ricerca dei sei gruppi più significativi tra gli individui campionati in tutte le razze dello studio. Si evince chiaramente come la BMI è nettamente separata dal resto delle razze formando un gruppo omogeneo con gli animali del Mozambico, che è evidentemente un derivato diretto della BMI. Nello studio sono incluse altre popolazioni presenti in Europa, Romania e Bulgaria, che formano anche chiaramente il loro cluster differenziato dalla BMI (Figure 4).
Figura 4 – Grafico a barre dove ogni individuo, rappresentato da una barra verticale, è colorato a seconda della sua appartenenza ad un determinato gruppo genetico (K=6). Analizzato e pubblicato per Colli et al., 2018.
Nell’anno 2021, viene pubblicato un altro articolo in cui è inclusa la Bufala Mediterranea Italiana (Noce et al., 2021): “Genetic Diversity of Bubalus bubalis in Germany and Global Relations of Its Genetic Background”. Nello studio sono state utilizzate informazioni genotipiche di animali provenienti da diversi allevamenti localizzati in Germania, Bulgaria, Romania ed Ungheria che sono state confrontate con genotipi di altre popolazioni mondiali disponibili in un database open-data. Lo studio ha identificato un alto grado di diversità genetica nel bufalo europeo e un nuovo gruppo di individui nella popolazione ungherese. Inoltre, è possibile osservare una netta separazione del bufalo europeo dalle altre popolazioni di bufali nel mondo (Figura 5).
La Figura 5 mostra i due gruppi principali della razza Mediterranea italiana e della razza Murrah. Nel primo gruppo, sono state unite le popolazioni italiane, del Mozambico e di tre allevamenti tedeschi. Nel secondo gruppo, le razze Murrah provenienti dal Pakistan e dall’India sono raggruppate con campioni provenienti dall’America Latina, dal Medio Oriente e dalla Bulgaria. In un altro gruppo spicca il bufalo Ungherese e Rumeno, mentre gli animali Sassoni sono raggruppati nel cluster Rumeno.
Figura 5 – Albero filogenetico che indica la distanza genetica di 22 popolazioni di bufali, analizzato e pubblicato in parte (Colli et al., 2018 e Deng et al., 2019).
La Figura 6 mostra invece la struttura genetica relativa esclusivamente alle popolazioni europee. Attraverso l’analisi di admixture vengono identificati un numero ottimale di 6 cluster (K), indicati ciascuno con un colore diverso. Sull’asse orizzontale sono riportate le popolazioni dalle quali provenivano i diversi campioni: è piuttosto evidente come si distinguano e separino tre componenti principali e ben definite (Italia, Ungheria e Bulgaria) e due componenti più variabili (Germania e Romania).
Figura 6 – Analisi de ADMIXTURE esclusivamente nelle popolazioni di bufali europee; K = 6 era la soluzione migliore per l’errore di convalida incrociato più basso riportato (CV = 0,59712).
Analizzando i diversi risultati raggiunti da Noce et al, quando sono inclusi campioni provenienti dai soli cinque paesi europei, si possono distinguere chiaramente quattro gruppi genetici, che rappresentano la popolazione bulgara, ungherese, rumena ed italiana. La popolazione tedesca ha mostrato un corredo molto misto ed è situata geneticamente tra la popolazione italiana e quella rumena. In particolare, gli animali della popolazione rumena appaiono sempre chiaramente separati, ma non è ancora ben chiara la loro origine. Secondo Borghese, si tratta di un tipo mediterraneo adattato al clima freddo e all’ambiente locale classificato come una razza dei Carpazi mediterranei (Borghese, 2005). Questo lavoro mirava a contribuire a una migliore comprensione delle popolazioni di bufali dell’Europa centrale e sudorientale da una prospettiva genomica. Sono state definite le caratteristiche genetiche delle popolazioni di bufali europee in Romania, Bulgaria e Ungheria. Insieme alla razza mediterranea italiana, queste popolazioni contribuiscono all’elevato grado di diversità genetica dei bufali europei (Noce et al., 2021).
In particolare, è stato possibile avere prove genetiche per distinguere una razza ungherese nuova o originale, caratterizzata dalla massima uniformità e senza chiare relazioni con qualsiasi altro genotipo descritto finora. Questa nuova identità genetica trovata nei bufali ungheresi aggiunge quindi un’ulteriore razza specifica alla diversità genetica esistente in Europa, che finora consisteva solo nella genetica italiana mediterranea, bulgara Murrah e rumena.
L’ultimo lavoro approfondito è “The distribution of runs of homozygosity in the genome of river and swamp buffaloes reveals a history of adaptation, migration and crossbred events”, pubblicato da Macciotta et al., (2021). Anche questo studio ha fatto ricorso ai pannelli di marcatori ad alta densità focalizzandosi sulla struttura genomica della popolazione genomica e sul livello di consanguineità (Macciotta et al., 2021). In questo caso è stata utilizzata una metodologia molto innovativa, basata sulle cosiddette ROH o runs of homozygosity. Le ROH sono regioni del cromosoma dell’animale costituite da gruppi di marcatori continui e con genotipo omozigote (Broman and Weber, 1999). L’analisi delle ROH nel genoma dei due tipi di bufalo d’acqua ha permesso la loro caratterizzazione genomica in maniera molto più precisa ed è in grado di evidenziare le differenze tra i tipi di bufalo e tra le razze (Macciotta et al., 2021).
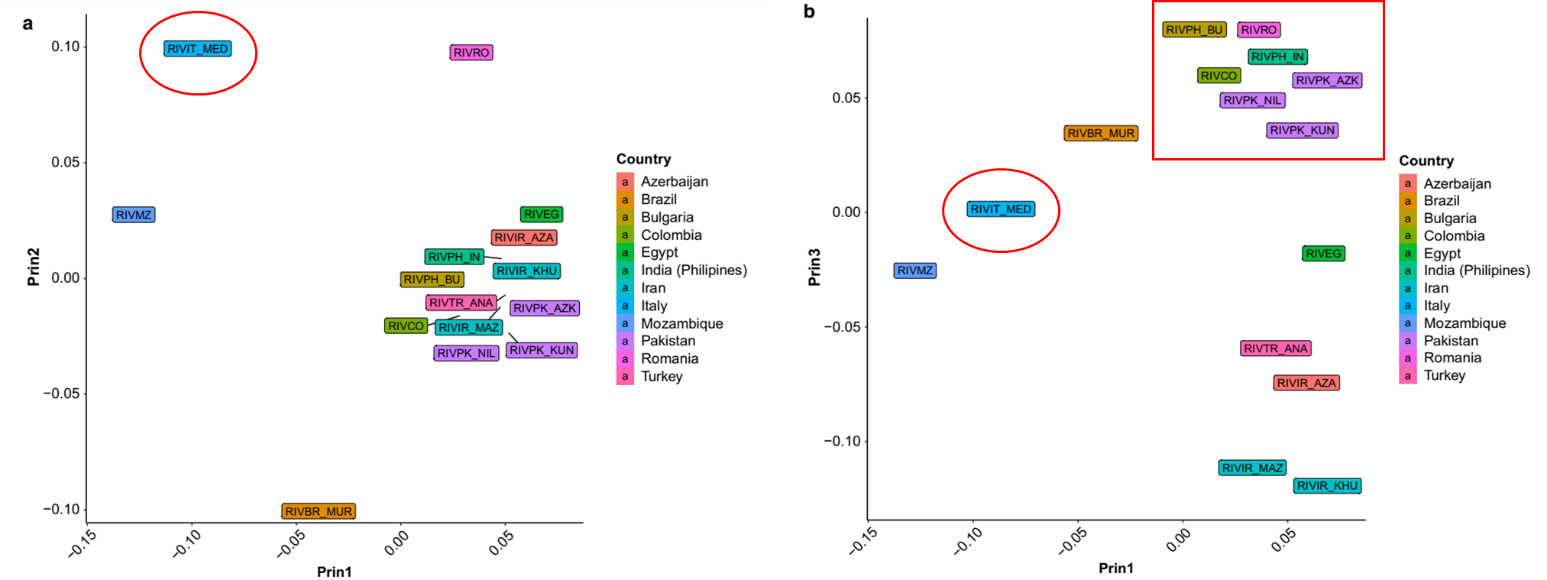
La Figura 7 mostra il raggruppamento tra le razze di bufalo di fiume, che segue in parte una distribuzione geografica, con orientamento Est-Ovest (Figura 7a), e che raggruppa le razze dei paesi dell’Est e del Medio Oriente nella parte destra della cornice e le due razze mediterranee, italiana (circolo rosso) e mozambicana, rispettivamente in alto a sinistra.
Figura 7 – Matrice di relazione genomica basata su ROH in bufali di fiume ottenuta attraverso l’analisi delle componenti principali. Prime due (a), e terza e seconda (b). Colori diversi indicano diversi paesi di origine.
La distribuzione delle razze di bufali di fiume non segue uno schema geografico rigoroso. Le Figure 7a, b mostrano che il bufalo di fiume iraniano, turco ed egiziano sono raggruppati, in base alla loro vicinanza geografica, mentre le razze provenienti da Italia, Mozambico e, in misura minore, Romania e Brasile sono separate dalle altre. Ciò può essere spiegato dal fatto che i bufali di fiume dall’Italia sono stati esportati in Mozambico, Brasile (Cockrill, 1974) e Romania, e quindi queste popolazioni condividono un background genomico “mediterraneo” comune. Interessante è la relativa vicinanza tra il bufalo rumeno e il bufalo bulgaro Murrah (Fig. 7b). La razza bulgara ha avuto origine da incroci tra animali autoctoni mediterranei e bufali indiani Murrah importati (Colli et al., 2018). Sono stati segnalati anche ulteriori incroci con bufali rumeni (Martikainen et al., 2018). Pertanto, la prima componente principale (Fig. 7a) potrebbe essere interpretata come un indice del contenuto “mediterraneo” del genoma, mentre la seconda dimensione (Fig. 7b) è un indice della transizione tra i genomi del Bufalo Mediterraneo e Murrah.
Considerazioni finali
Il fenotipo di un individuo ha da sempre rappresentato uno dei metodi più semplici e diretti per “raggruppare” gli animali. Dal fenotipo si parte anche per fare selezione, come Robert Bakewell intuì nel 18esimo secolo. Dietro al fenotipo c’è però il genoma ed oggi è molto più facile ed immediato di prima capire come si relazionano razze o sub-specie diverse e come utilizzare queste informazioni per “fare” selezione in modo corretto, funzionale ed efficiente.
Autori
A cura di M. Gómez1, G. Zullo1 e S. Biffani2
1 ANASB – Associazione Nazionale Specie Bufalina, Caserta
2 IBBA – Istituto di Biologia e Biotecnologia Agraria – Consiglio Nazionale delle Ricerche, Milano
Bibliografia
Borghese, A. 2005. Buffalo production and research. FAO Ed. REU Tech. Ser. 67, 1–315.
Borghese, A. and B. Moioli. 2016. Buffalo: Mediterranean Region. Reference Module in Food Science:B978-970-908-100596-100595.121232-100598.
Broman, K. W. and J. L. Weber. 1999. Long homozygous chromosomal segments in reference families from the centre d’Etude du polymorphisme humain. Am. J. Hum. Genet. 65(6):1493-1500.
Cockrill, W. 1974. The husbandry and health of the domestic buffalo. Rome: Food and Agriculture Organization of the United Nations.
Colli, L., M. Milanesi, E. Vajana, D. Iamartino, L. Bomba, F. Puglisi, M. Del Corvo, E. L. Nicolazzi, S. S. E. Ahmed, J. R. V. Herrera, L. Cruz, S. Zhang, A. Liang, G. Hua, L. Yang, X. Hao, F. Zuo, S.-J. Lai, S. Wang, R. Liu, Y. Gong, M. Mokhber, Y. Mao, F. Guan, A. Vlaic, B. Vlaic, L. Ramunno, G. Cosenza, A. Ahmad, I. Soysal, E. Ö. Ünal, M. Ketudat-Cairns, J. F. Garcia, Y. T. Utsunomiya, P. S. Baruselli, M. E. J. Amaral, R. Parnpai, M. G. Drummond, P. Galbusera, J. Burton, E. Hoal, Y. Yusnizar, C. Sumantri, B. Moioli, A. Valentini, A. Stella, J. L. Williams, and P. Ajmone-Marsan. 2018. New Insights on Water Buffalo Genomic Diversity and Post-Domestication Migration Routes From Medium Density SNP Chip Data. Frontiers in Genetics 9:53.
FAO. Food and Agriculture Organization (FAO). Live Animals. (2020). Available online at: http://www.fao.org/faostat/en/#data/QA [Accessed September 28, 2020).
Iamartino, D., Nicolazzi, E. L., Van Tassell, C. P., Reecy, J. M., Fritz-Waters, E. R., Koltes, J. E., Biffani, S. et al. (2017). Design and validation of a 90K SNP genotyping assay for the water buffalo (Bubalus bubalis). PLoS One 12:e0185220. doi: 10.1371/ journal.pone.0185220
Li, M., F.-u. Hassan, Y. Guo, Z. Tang, X. Liang, F. Xie, L. Peng, and C. Yang. 2020. Seasonal Dynamics of Physiological, Oxidative and Metabolic Responses in Non-lactating Nili-Ravi Buffaloes Under Hot and Humid Climate. Frontiers in Veterinary Science 7:622.
Macciotta, N. P. P., L. Colli, A. Cesarani, P. Ajmone-Marsan, W. Y. Low, R. Tearle, and J. L. Williams. 2021. The distribution of runs of homozygosity in the genome of river and swamp buffaloes reveals a history of adaptation, migration and crossbred events. Genet Sel Evol 53(1):20.
Martikainen, K., A. Sironen, and P. Uimari. 2018. Estimation of intrachromosomal inbreeding depression on female fertility using runs of homozygosity in Finnish Ayrshire cattle. J. Dairy Sci. 101(12):11097-11107.
Minervino, A. H. H., M. Zava, D. Vecchio, and A. Borghese. 2020. Bubalus bubalis: A Short Story. Frontiers in veterinary science 7:570413-570413.
Moioli, B. and A. Borghese. 2005. Buffalo breeds and management systems. In: Borghese A, editor. Buffalo Production and Research. Rome: FAO. p. 51–108.
Mokhber, M., M. M. Shahrbabak, M. Sadeghi, H. M. Shahrbabak, A. Stella, E. Nicolzzi, and J. L. Williams. 2019. Study of whole genome linkage disequilibrium patterns of Iranian water buffalo breeds using the Axiom Buffalo Genotyping 90K Array. PLOS ONE 14(5):e0217687.
Noce, A., S. Qanbari, R. González-Prendes, J. Brenmoehl, M. G. Luigi-Sierra, M. Theerkorn, M.-A. Fiege, H. Pilz, A. Bota, L. Vidu, C. Horwath, L. Haraszthy, P. Penchev, Y. Ilieva, T. Peeva, W. Lüpcke, R. Krawczynski, K. Wimmers, M. Thiele, and A. Hoeflich. 2021. Genetic Diversity of Bubalus bubalis in Germany and Global Relations of Its Genetic Background. Frontiers in Genetics 11:1664.
Yadav, A. K., J. Singh, and S. K. Yadav. 2017. Characteristic features of registered indigenous buffalo breeds of India: a review. Int. J. Pure App. Biosci 5(4):825-831.










































































Scrivi un commento
Devi accedere, per commentare.